Przeczytaj
Regulacja ekspresji genów to niezmiernie ważny i precyzyjny mechanizm, który umożliwia komórkom uruchamianie tylko tych genów, które są im potrzebne do realizacji potrzeb życiowych. Szczególnie widoczne jest to w przypadku komórek organizmów wielokomórkowych, które ulegają specjalizacji i pewne swoje funkcje ograniczają, by skuteczniej realizować inne. Każda z komórek organizmu wielokomórkowego, w trakcie rozwoju płodowego, powstaje z komórek totipotencjalnychtotipotencjalnych. Ich specjalizacja polega więc między innymi na stymulacji aktywności genów kluczowych dla danego typu komórek oraz ograniczaniu aktywności zbędnych genów. Dzieje się to za pomocą mechanizmów regulacji ich ekspresji.
W komórkach prokariotycznych również istnieją mechanizmy regulacji genów. Bakterie potrafią w pewnym stopniu dostosowywać metabolizm do warunków środowiska oraz zapotrzebowania metabolicznego. Dzieje się tak dzięki możliwości włączania i wyłączania określonych genów. Charakterystyczną cechą komórek bakterii jest grupowanie głównych genów jednego szlaku metabolicznego w operonyoperony. Dzięki skupieniu genów w operon komórka prokariotyczna może uruchomić lub zahamować ekspresję całej grupy genów naraz, w zależności od zapotrzebowania.
Zasady regulacji ekspresji genów u prokariontów
Model operonu został odkryty przez François Jacoba i Jacques’a Monoda w 1961 r. W jego skład, oprócz genów konkretnego szlaku metabolicznego, wchodzą dwa ważne odcinki DNA, pełniące nadrzędną kontrolną funkcję wobec pozostałych genów. Pierwszym z nich jest promotorpromotor, czyli sekwencja DNA, którą rozpoznaje polimeraza RNApolimeraza RNA. Drugi odcinek, czyli operatoroperator, jest sekwencją specyficzną wyłącznie dla struktury operonu. Występuje w pobliżu promotora i to do niej przyłącza się represorrepresor, czyli białko, które wpływa na zahamowanie transkrypcjitranskrypcji operonu. Więcej informacji na ten temat znajdziesz w e‑materiale: Struktura i sposoby regulacji operonów prokariotycznych.
Operon może ulegać regulacji negatywnej lub pozytywnej. Regulacja negatywna związana jest z działaniem represora, który łączy się z operatorem i blokuje działanie operonu. Uniemożliwia to przyłączenie się polimerazy RNA i rozpoczęcie transkrypcji. Przykładem operonu o regulacji negatywnej jest operon tryptofanowy, który zawiera geny kodujące enzymy szlaku produkcji tryptofanu. W obecności tryptofanu produkowany w formie nieaktywnej represor ulega aktywacji i uniemożliwia transkrypcję genów operonu, zapobiegając nadmiernej produkcji tego aminokwasu. W warunkach niedoboru tryptofanu represor pozostaje nieaktywny, co odblokowuje ekspresję genów operonu i syntezę tego aminokwasu. Dokładny opis tego mechanizmu znajduje się w e‑materiale: Operony hamowane – operon tryptofanowy.
Regulacja pozytywna natomiast polega na aktywacji ekspresji genów operonu. Dzieje się tak w wyniku wiązania aktywatoraaktywatora w specjalnym miejscu przed promotorem, który ułatwia przyłączenie polimerazy do DNA i rozpoczęcie transkrypcji. Regulacji pozytywnej podlega np. operon laktozowy, zawierający geny szlaku rozkładu laktozy. W obecności laktozy i przy niewielkim stężeniu glukozy aktywator przyłącza się do określonej sekwencji w DNA, ułatwiając przyłączenie polimerazy RNA, co umożliwia ekspresję genów operonu z dużą wydajnością. Co ciekawe, operon laktozowy podlega również regulacji negatywnej. Więcej informacji na ten temat znajdziesz w e‑materiale: „Operony indukowane – operon laktozowy”.
Zasady regulacji ekspresji genów u eukariontów
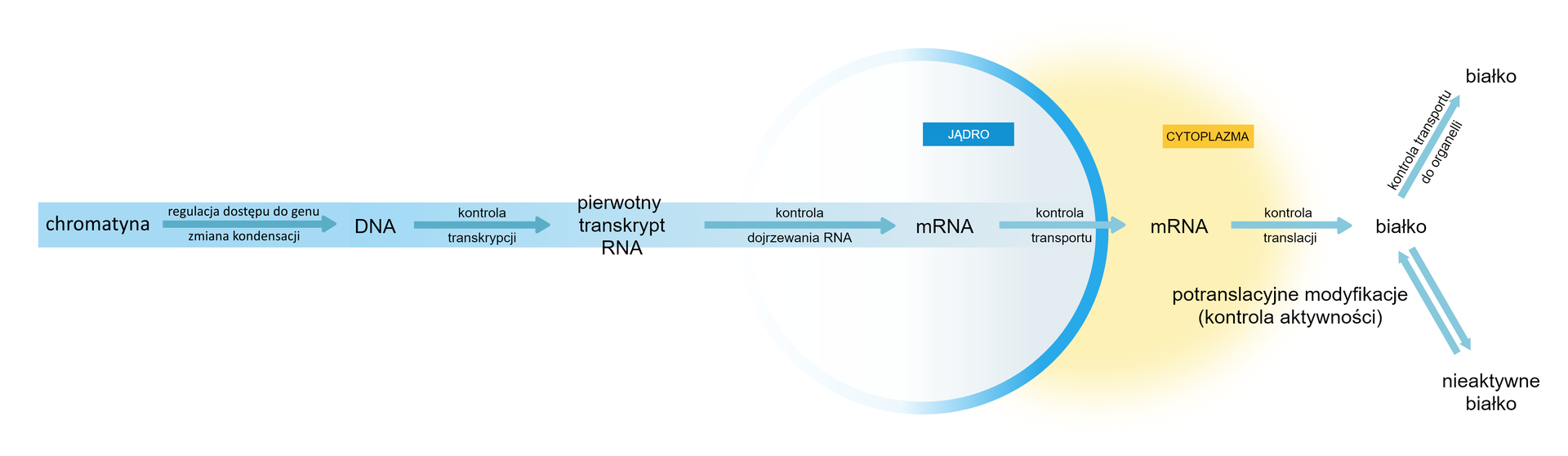
Komórki eukariotyczne mają znacznie bardziej skomplikowane mechanizmy regulacji genów niż bakterie. Brak u nich operonów, a ekspresja każdego genu zachodzi osobno. Jest to związane z budową komórek eukariotycznych (obecnością obłonionego jądra i rozdzielenia przestrzennego poszczególnych etapów ekspresji), a także z wysoką specjalizacją w pełnieniu funkcji w wielokomórkowym organizmie. Proces ekspresji genów jest bardziej skomplikowany niż u prokariontów, a regulacja odbywa się w kilku etapach. Aby geny mogły zostać aktywowane, najpierw chromatyna musi ulec rozluźnieniu. Jest to tzw. regulacja dostępu do genu. Wiąże się ona z modyfikacją chemiczną histonów, co powoduje zmianę kondensacji chromatyny i ułatwia dostęp do konkretnego odcinka DNA. Proces ten jest odwracalny.
Kolejny mechanizm regulacji dotyczy rozpoczęcia procesu transkrypcji. Wiąże się on z obecnością czynników transkrypcyjnych, których przyłączenie do DNA ułatwia związanie polimerazy RNA. U eukariontów występują bardzo liczne odcinki regulatorowe genów oraz przyłączane czynniki transkrypcyjne. Wiele czynników jest specyficznych dla danego typu komórek i nie występuje w komórkach innych rodzajów. Geny mogą być aktywowane osobno lub w sposób skoordynowany (szczególnie w przypadku genów jednego szlaku metabolicznego).
Regulacja odbywa się również na poziomie procesu translacjitranslacji. Produkcja białek na podstawie matrycowego RNA (mRNA)matrycowego RNA (mRNA) może być aktywowana, np. przez syntezę większej ilości rybosomów. Może być także hamowana, np. przez degradację powstałego mRNA czy terminację translacji i degradację powstałego peptydu. Co więcej, ekspresja jednego genu umożliwia czasami wytworzenie białek różnego typu – w tym przypadku regulacja odbywa się podczas wcześniejszego etapu: modyfikacji potranskrypcyjnej pre‑mRNA. Matrycowy RNA może ulegać tzw. alternatywnemu składaniu, w którym wykorzystywane są tylko niektóre odcinki kodujące pre‑mRNA.
Ostatni mechanizm regulacji związany jest z białkami regulatorowymi oraz miRNAmiRNA (mikroRNA), czyli krótkimi, jednoniciowymi, niekodującymi cząsteczkami RNA. Odgrywają one bardzo ważną rolę w blokowaniu translacji mRNA, a nawet powodują jego rozpad. Z kolei białka regulatorowe mają zdolność aktywacji lub blokowania translacji, przez odpowiednie łączenie się z mRNA.
Słownik
białko przyłączające się do DNA, które wiąże się z odpowiednim miejscem operonu i indukuje lub zwiększa transkrypcję jego genów
(ang. micro RNA) mały niekodujący RNA, wyciszający ekspresję genów
(ang. messenger RNA) matrycowy kwas rybonukleinowy; w procesie translacji stanowi matrycę zawierającą informację o sekwencji aminokwasów w białku
sekwencja DNA w operonie regulująca aktywność genów struktury, do której wiąże się białko regulatorowe (represor)
specyficzna i powszechna u bakterii strukturalna i funkcjonalna jednostka genomu; zawiera od dwóch do kilku kolejno ułożonych genów strukturalnych, które kodują enzymy jednego szlaku metabolicznego, oraz położone bezpośrednio przed genami sekwencje operatora i promotora, które kontrolują ich aktywność transkrypcyjną; termin operon wprowadzili do genetyki w 1961 roku François Jacob i Jacques Monod, odkrywcy mechanizmu jego regulacji
enzym odpowiadający za przeprowadzenie procesu transkrypcji
odcinek DNA zawierający sekwencje rozpoznawane przez polimerazę RNA zależną od DNA
białko regulatorowe wiążące się z określoną sekwencją DNA w operatorze i hamujące proces transkrypcji genów; represor uniemożliwia przyłączanie się polimerazy RNA do promotora
proces syntezy RNA, podczas którego na matrycy DNA jest syntetyzowana komplementarna nić RNA
drugi etap ekspresji genu, podczas którego na podstawie informacji zapisanej w nici mRNA jest syntetyzowany polipeptyd
komórki zdolne do wielokierunkowego różnicowania się, pojawiające się na wczesnych etapach rozwoju zarodkowego (bruzdkowania)