Przeczytaj
Drzewo filogenetyczne
Do prezentacji wyników badań ewolucji, czy to na podstawie materiału genetycznego żyjących organizmów, czy też z użyciem materiału kopalnego, bardzo często wykorzystuje się drzewa filogenetycznedrzewa filogenetyczne i kladogramykladogramy. Na ich podstawie wyróżnia się grupy systematyczne.
Drzewo filogenetyczne, nazywane również filogramem, to ukorzeniony diagram przedstawiający relacje ewolucyjne między organizmami. Można je skalibrować względem czasu geologicznego i wskazać kierunek ewolucji. Diagram nieukorzeniony to kladogramkladogram, natomiast grupa organizmów mających wspólnego przodka określana jest jako kladklad. W analizie kladystycznejanalizie kladystycznej drzewo filogenetyczne zawiera informacje z kladogramu uzupełnione o sekwencje przodków i potomków.
Należy również pamiętać, że drzewo filogenetyczne może reprezentować pełną różnorodność życia wywodzącego się od wspólnego przodka (tak jak drzewo po lewej stronie grafiki) lub pojedynczą gałąź pełnego drzewa życia, takiego jak linie kręgowców, grzybów, paprotników (tak jak po prawej stronie grafiki).
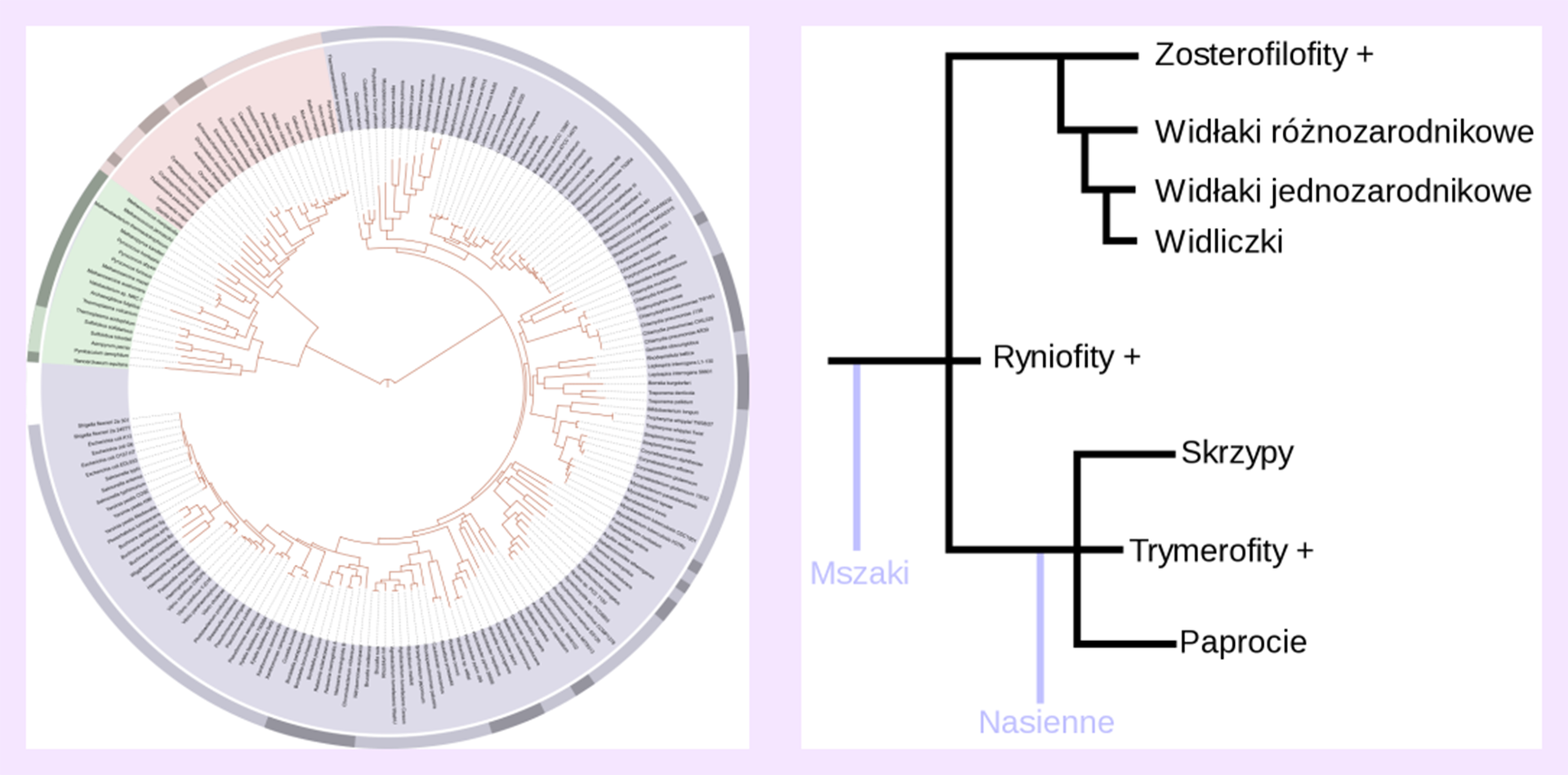
Po prawej stronie znajduje się drzewo filogenetyczne paprotników – widać odnogi do mszaków i nasiennych. Znakiem "+" oznaczono gatunki wymarłe.
Drzewa filogenetyczne tworzy się w celu określenia filogenezy organizmów poprzez badanie ich chronologicznej drogi rozwojowej i stopnia pokrewieństwa między organizmami żyjącymi współcześnie oraz kopalnymi.
Drzewa filogenetyczne to hipotezy, a nie ostateczne fakty.
Budowa drzewa filogenetycznego
Budowa drzew filogenetycznych opiera się na informacjach dotyczących analizowanych gatunków – takich jak cechy fizyczne i sekwencje DNA ich genów.
Korzeń drzewa reprezentuje wspólnego przodka, natomiast wzór rozgałęzień odzwierciedla sposób, w jaki gatunki lub inne grupy wyewoluowały z szeregu wspólnych przodków. Długość gałęzi i kąty zawarte między nimi określają tempo zmian w potomnych liniach. Linie, określane jako gałęzie drzewa, kończą się liśćmi symbolizującymi organizmy współcześnie żyjące. Wymarłe grupy ulokowane są poniżej.
Każdy punkt rozgałęzienia (zwany także węzłem wewnętrznym) reprezentuje zdarzenie dywergencji, czyli rozdzielenie pojedynczej grupy na dwie grupy podrzędne.
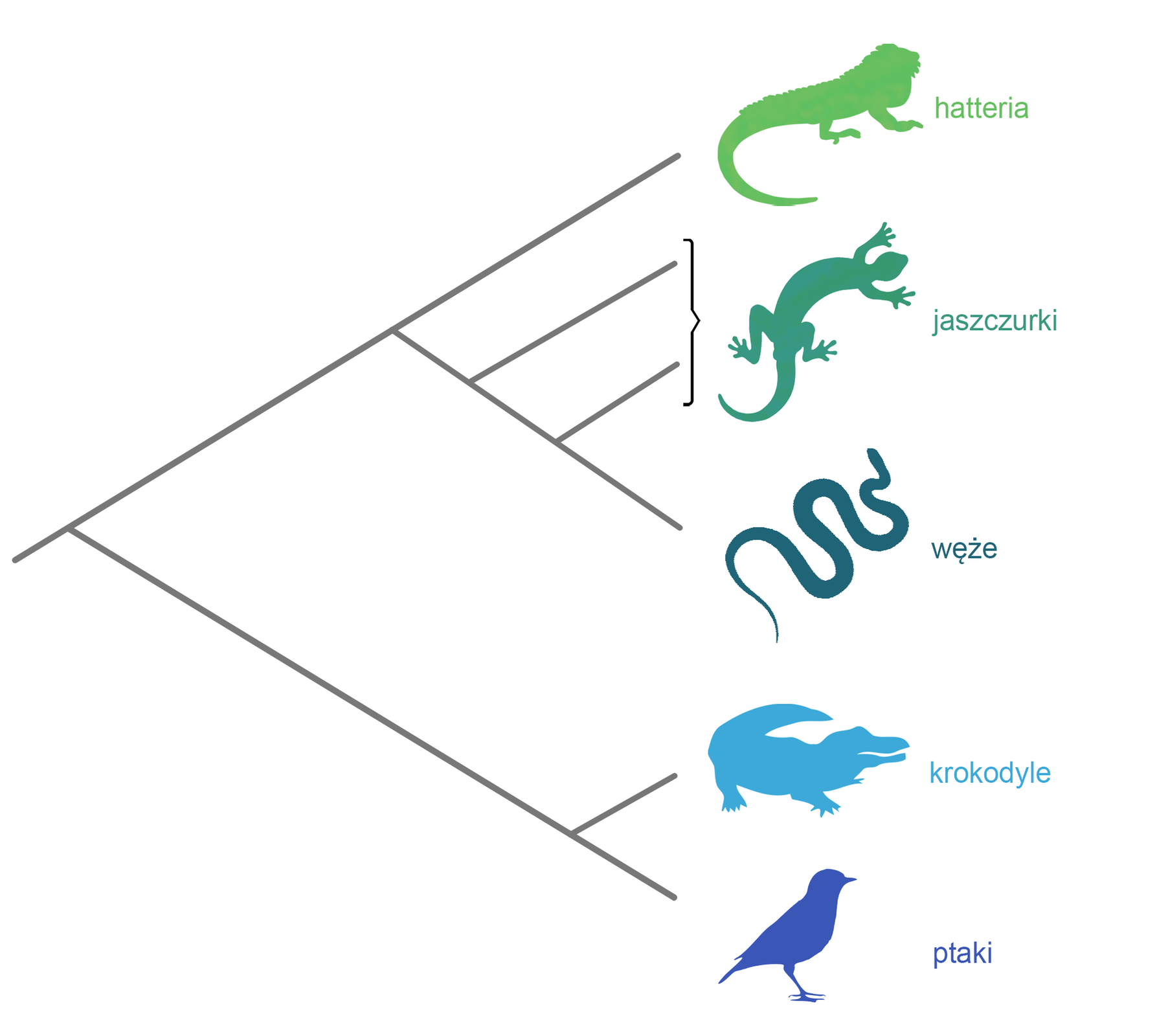
Na diagramie dwa gatunki są bliżej spokrewnione, jeśli mają niedawnego wspólnego przodka i mniej spokrewnione, jeśli mają starszego wspólnego przodka. Im mniej węzłów oddziela od siebie dwa taksonytaksony, tym bliżej są one ze sobą spokrewnione. Na przykładzie drzewa filogenetycznego gadów i ptaków można ustalić, które z tych grup organizmów są ze sobą najbliżej spokrewnione. Z tego drzewa wynika, że ptaki oraz krokodyle miały wspólnego przodka, a krokodyle są bliżej spokrewnione z ptakami niż z innymi gadami.
Czytanie drzew filogenetycznych
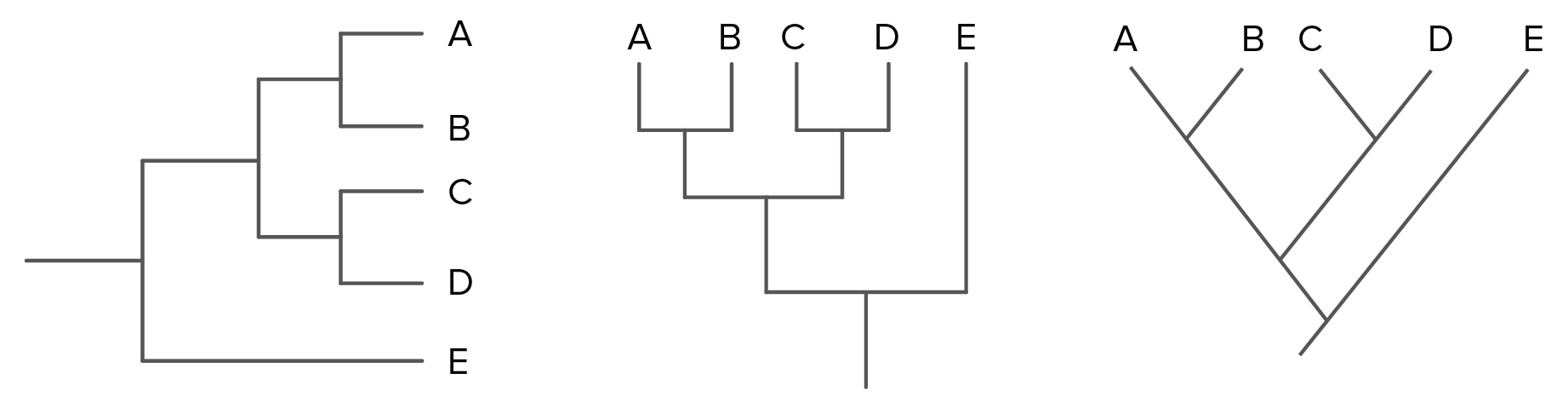
Drzewa filogenetyczne mogą przyjmować różne formy. Przedstawione poniżej drzewa reprezentują identyczne relacje między gatunkami A, B, C, D i E.
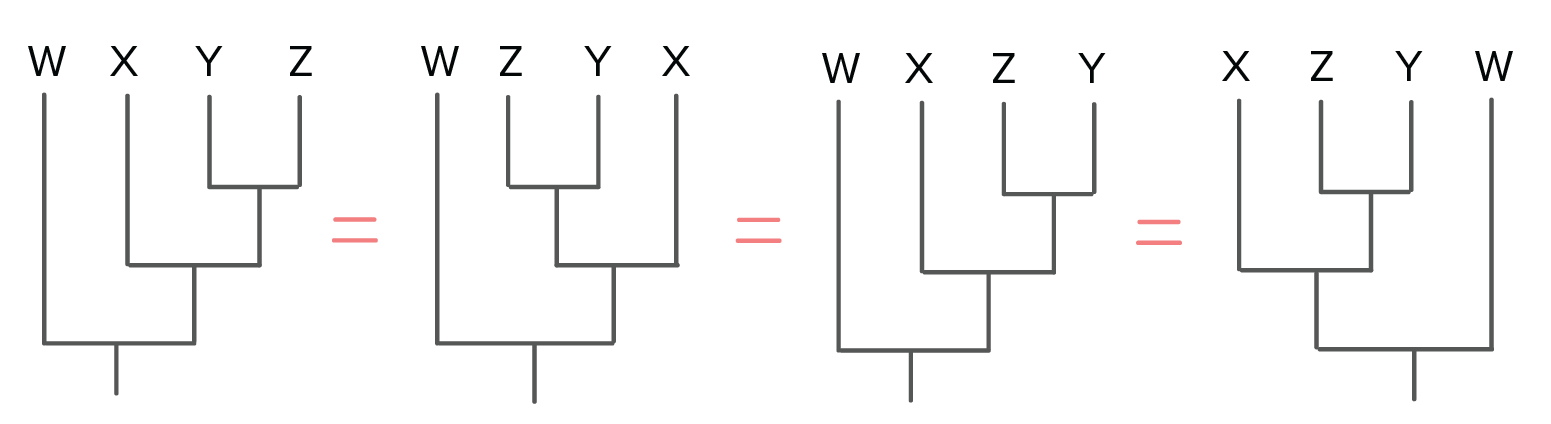
Drzewa filogenetyczne można również rysować w różnych, równoważnych stylach. Obracanie drzewa w punktach rozgałęzień nie zmienia informacji, które ono zawiera.
Tworzenie drzew filogenetycznych
Podczas tworzenia drzew filogenetycznych porównuje się i analizuje wiele cech gatunków (lub innych grup). Cechy te mogą obejmować morfologię zewnętrzną (wygląd, kształt, wielkość), anatomię wewnętrzną, zachowania, szlaki biochemiczne, sekwencje DNA, a nawet charakterystykę skamieniałości.
Występowanie określonej cechy u przedstawicieli danej linii ewolucyjnej oraz u ich przodków to plezjomorfiaplezjomorfia. Przykładem plezjomorfii jest obecność pięciu palców na dłoniach i stopach ludzi – cecha ta występuje również u płazów i gadów.
Ważnym aspektem ustalania pokrewieństwa między organizmami było wyodrębnianie taksonów na podstawie cech pochodnych (apomorficznych), czyli takich, które pojawiły się na drodze ewolucji w danej grupie i zostały odziedziczone przez gatunki potomne.
SynapomorfiaSynapomorfia (cecha zaawansowana) to apomorfia dzielona przez co najmniej dwa taksony, najprawdopodobniej powstała u ich najnowszego wspólnego przodka. Można ją wyróżnić u wszystkich przedstawicieli danej grupy filogenetycznej. Przykładem synapomorfii jest obecność cech pochodnych takich jak wyprostowany chód, futro i gruczoły sutkowe u ssaków. Cechy te nie występują u innych kręgowców, takich jak płazy i gady, które zachowały przejęte po przodkach cechy chodu rozwlekłego i braku futra. Zatem każda z tych cech pochodnych jest apomorfią poszczególnych gatunków ssaków i synapomorfią ssaków, której nie mają inne kręgowce.
AutapomorfiąAutapomorfią nazywa się z kolei cechę, która występuje tylko w jednej grupie filogenetycznej. Jest nią np. obecność sierści i gruczołów mlekowych u ssaków.
Współcześnie znaczną część analizy pokrewieństw między taksonami wykonują komputery. Zadaniem badacza jest dobranie odpowiedniej metody do posiadanych danych, a następnie interpretacja wyników. Samych obliczeń dokonują programy komputerowe. Wynikiem takich obliczeń jest kladogram.
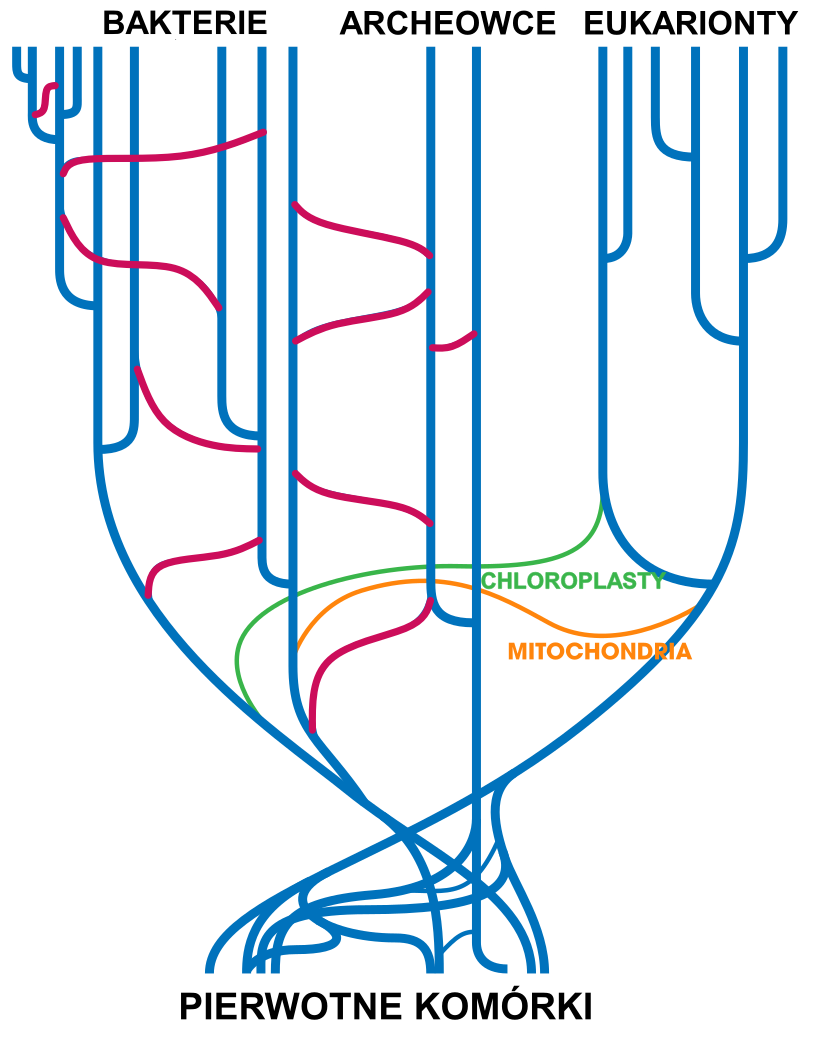
Kiedy opracowano dokładniejsze metody analizy sekwencji DNA, okazało się, że część wyników wcześniejszych badań RNA wykazuje różnice. Wynika to ze zjawiska horyzontalnego transferu genówhoryzontalnego transferu genów, związanego z pozyskaniem genu od innego organizmu (przekazaniem genu przez organizm niebędący przodkiem biorcy genu). Horyzontalny transfer genów jest głównym mechanizmem rozprzestrzeniania się m.in. oporności na antybiotyki u bakterii.
Przełomem w badaniach molekularnych, wykorzystywanych również w analizach filogenetycznych, było sekwencjonowanie metodą SangeraSangera.
Więcej informacji na temat metody Sangera w materiale pt. „Sekwencjonowanie DNA”.
Jednostki taksonomiczne
W oparciu o drzewa filogenetyczne można wyróżniać jednostki taksonomiczne (systematyczne).
Grupa monofiletyczna to grupa obejmująca wszystkich potomków ostatniego wspólnego przodka. Grupa ta nazywana jest również klademkladem.
Grupa parafiletyczna nie obejmuje wszystkich potomków ostatniego wspólnego przodka.
Grupa polifiletyczna łączy taksony mogące należeć do bardzo różnych, niezależnych grup monofiletycznych, o różnym stopniu pokrewieństwa, ale wykazujące podobieństwa budowy lub fizjologii.
Filogeneza zwierząt
Pojęcie filogenezyfilogenezy wprowadził w 1866 r. Ernst HaeckelErnst Haeckel, definiując ją jako „historię rozwoju paleontologicznego gatunków”. Współcześnie termin ten rozumiany jest jako całokształt ewolucyjnego przekształcania się budowy i funkcji następujących po sobie generacji różnych grup organizmów (gatunków).
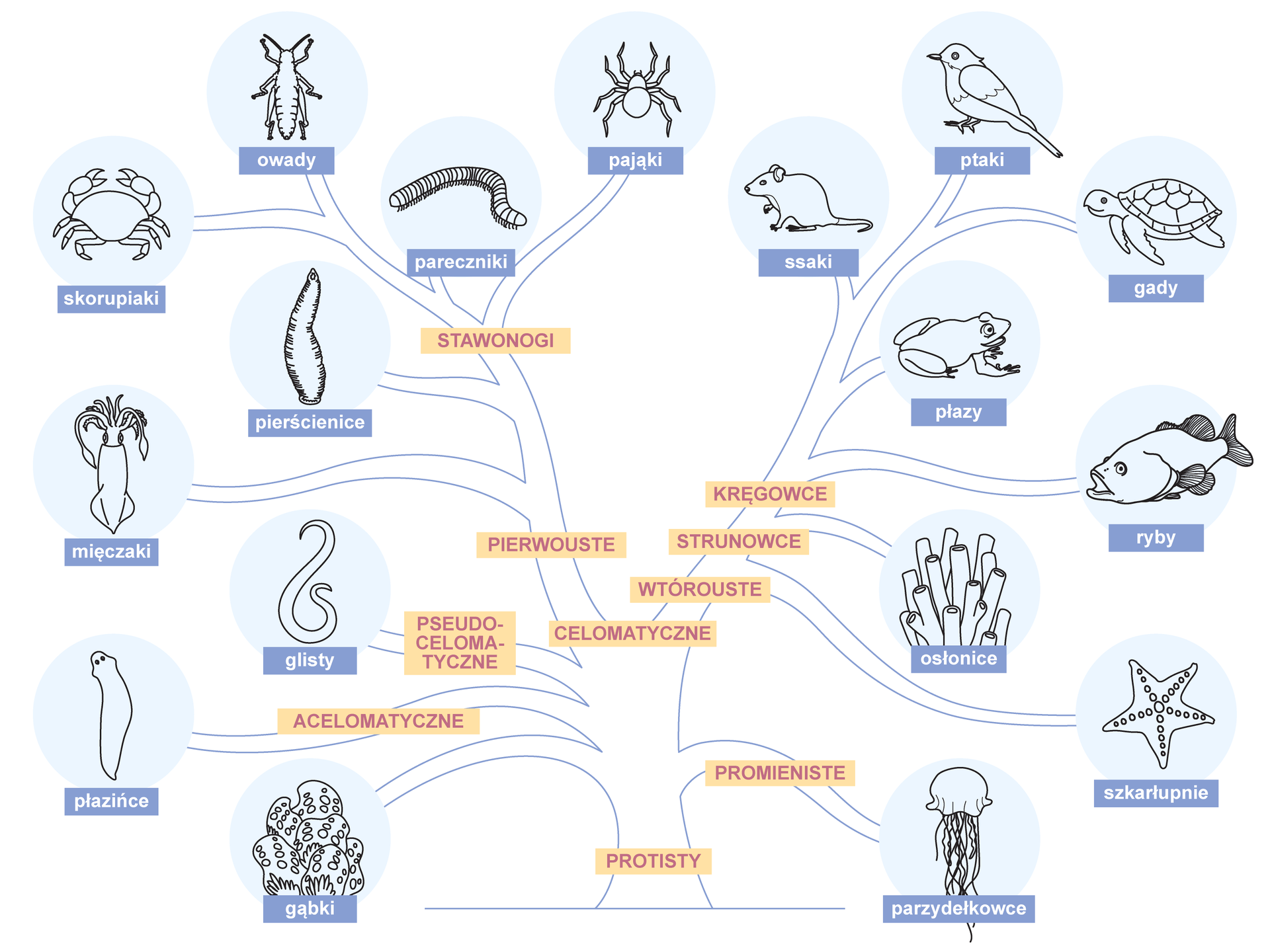
Początki filogenezy zwierząt sięgają prekambru. Przodkami zwierząt wielokomórkowych były wiciowce. Najstarsze znane skamieniałości zwierzęce pochodzą sprzed 700 mln lat i należą do licznie występujących w kambrze priapulidów (Priapuloidea), od których wyprowadza się robaki obłe. Do robaków obłych zaliczane są m.in. glisty, będące przedstawicielami nicieni. Na przełomie prekambru i kambru pojawiły się pierwsze mięczaki, we wczesnym kambrze – morskie pratchawce i najpierwotniejsze stawonogi. W środkowym kambrze dominowały trylobity oraz skorupiaki, pokrewne dzisiejszym cienkopancerzowcom (Leptostraca). W starszym paleozoiku dokonało się wyodrębnienie głównych pni rozwojowych szczękoczułkowców (do których należą pajęczaki) i skorupiaków. Klad, który obejmuje skorupiaki oraz owady (należące do sześcionogów), nosi nazwę Pancrustacea. Początki strunowców nie są znane, lecz dzięki szczątkom kopalnym wyodrębnienie szkarłupni datuje się na dolny kambr.
Klad, do którego należą organizmy strunowe (Chordata) oraz szkarłupnie, to wtórouste. Pierwszą grupą strunowców o znacznym zróżnicowaniu były konodonty, a współcześnie występującym podtypem strunowców są osłonice (Tunicata). W środkowym ordowiku pojawiły się pancerne bezszczękowce. Z jednej ich gałęzi wyodrębniły się akantody (o dwóch parach płetw parzystych), od których wywodzą się zapewne pozostałe grupy ryb. W późnym dewonie z ryb trzonopłetwych wyodrębniły się płazy. Najstarszym znanym czworonogiem jest ichtiostega, znaleziona na Grenlandii.
Z dominujących pod koniec paleozoiku i w triasie płazów tarczogłowych wyodrębniły się płazy bezogonowe i ogoniaste. Nie jest znane ogniwo pośrednie między rozwijającymi się w wodzie płazami a przechodzącymi rozwój na lądzie gadami. Najstarszymi znanymi gadami były antrakozaury. Z końcem permu pojawiły się gady naczelne, w późnym triasie – żółwie. Od tego też czasu znane są dinozaury i gady latające, właściwe jaszczurki – od jury, węże – od wczesnej kredy. Od drobnych, biegających dinozaurów pochodzą ptaki. Pierwszymi ptakami były późnojurajski archeopteryks i wczesnokredowy Sinornis. Z gadów ssakokształtnych na przełomie triasu i jury wyodrębniły się ssaki, których wielkie różnicowanie rozpoczęło się po wyginięciu dinozaurów, od początku trzeciorzędu. Różnicowanie się w gałęzi ewolucyjnej afrykańskich ssaków naczelnych doprowadziło ok. 4,4 mln lat temu do powstania dwunożnych australopiteków – przodków dzisiejszego Homo sapiens.
Korzeń oznacza wspólnego przodka dla wszystkich taksonów; gałąź obrazuje związki ewolucyjne między porównywanymi jednostkami taksonomicznymi, długość gałęzi reprezentuje liczbę zmian, które nastąpiły w danej linii ewolucyjnej, węzeł reprezentuje miejsce rozgałęzień jednostek taksonomicznych (populacji, organizmu, genu). Najczęściej jest to ostatni wspólny przodek grup pochodzących od węzła (miejsca rozgałęzienia). Natomiast liść przedstawia aktualnie analizowaną jednostkę taksonomiczną.
Rekonstrukcja drzew filogenetycznych
Rekonstrukcja drzew filogenetycznych opiera się na badaniach z zakresu biochemii i genetyki, które pozwalają z dużą dokładnością określić podobieństwa i różnice między poszczególnymi taksonami i gatunkami. Badania te obejmują m.in.:
określanie i porównywanie sekwencji aminokwasów tych samych białek występujących u różnych gatunków (np. cytochromu B i dehydrogenazy NADH);
określanie i porównywanie sekwencji nukleotydów z odpowiadających sobie genów u organizmów należących do różnych gatunków (najczęściej są to mitochondrialny DNA, rybosomalny DNA, cDNA, RNA).
Dowodami na istnienie powiązań ewolucyjnych między organizmami są również podobieństwa struktur i funkcji różnych związków występujących w komórce. Należą do nich m.in.:
taka sama struktura wielkocząsteczkowych związków organicznych (przede wszystkim białek i kwasów nukleinowych) wchodzących w skład wszystkich organizmów;
wykorzystywanie ATP jako podstawowego akumulatora i nośnika energii w komórkach wszystkich organizmów;
wytwarzanie przez organizmy większości zwierząt (od gąbek do ssaków) niektórych enzymów trawiennych, np. amylazy;
podobieństwo budowy chlorofilu (barwnika aktywnego podczas fotosyntezy) do budowy hemoglobiny (barwnika uczestniczącego w transporcie gazów oddechowych u zwierząt);
podobieństwo budowy niektórych istotnych dla funkcjonowania organizmu białek, np. cytochromu C;
podobieństwo niektórych hormonów zwierzęcych i ludzkich, np. insuliny ludzkiej i świńskiej;
podobieństwo budowy antygenów grupowych krwi u człowieka i małp człekokształtnych.
Słownik
metoda klasyfikacji organizmów na podstawie wspólnego pochodzenia ewolucyjnego
(gr. autos – ja, apo – z dala od i morphḗ – kształt) cecha unikatowa, występująca tylko u jednego gatunku bądź w jednej grupie filogenetycznej, niewystępująca u przodków
graficzne przedstawienie związków pokrewieństwa między taksonami i ich przeszłości ewolucyjnej; pień drzewa reprezentuje wspólnego przodka, rozgałęzienia zaś obrazują pokrewieństwo taksonów i ich czas powstania
(gr. phylḗ – plemię, ród i génesis – pochodzenie) rozwój rodowy organizmów; przebieg ewolucyjnego różnicowania organizmów jako rezultat wyodrębniania się nowych linii rozwojowych i wymierania innych
(1834–1919) niemiecki zoolog, ewolucjonista i filozof; w 1869 r. po raz pierwszy użył terminu „ekologia”, określającego badania nad zwierzętami oraz ich relacjami ze środowiskiem i wzajemnymi relacjami między organizmami; opracował pierwsze drzewo rodowe wszystkich organizmów, obejmujące również człowieka; ostatecznie sformułował tzw. prawo biogenetyczne, głoszące, że historia rozwoju organizmu (ontogeza) jest skróconym powtórzeniem jego historii ewolucyjnej (filogenezy)
zjawisko pozyskania genów od organizmu należącego do innego gatunku; proces wykorzystywany w warunkach laboratoryjnych do modyfikacji genetycznych
(gr. kládos – gałąź, pęd) grupa organizmów mająca wspólnego przodka oraz wszystkie organizmy wywodzące się z tej linii
graficzne przedstawienie pokrewieństwa grup organizmów bez uwzględnienia czasu pojawiania się nowych linii ewolucyjnych
(gr. kládos – gałąź, pęd) metoda klasyfikacji organizmów i ustalania ich historii ewolucyjnej (filogenetyka), sformułowana przez niemieckiego entomologa Williego Henniga
(gr. plesio − prawie i morphḗ – kształt) cecha prymitywna, występująca u filogenetycznie wcześniejszych przodków
(1918–2013) brytyjski biochemik; dwukrotny laureat Nagrody Nobla; w 1958 r. otrzymał Nagrodę Nobla w dziedzinie chemii za pracę nad strukturą białek, zwłaszcza insuliny; w 1980 r. Nagroda Nobla w dziedzinie chemii została podzielona: połowę przyznano Paulowi Bergowi za badania biochemii kwasów nukleinowych, zwłaszcza technik rekombinacyjnych, drugą połowę przyznano wspólnie Walterowi Gilbertowi i Frederickowi Sangerowi za wkład w określenie sekwencji zasad w kwasach nukleinowych
cecha zaawansowana, która występuje u wszystkich przedstawicieli danej grupy filogenetycznej, w kladystyce synapomorfia jest synonimem homologii
(gr. táksis – układ, porządek) jednostka systematyczna organizmów; taksony wyodrębnia się według przyjętych zasad, a następnie szereguje w porządku hierarchicznym (od najwyższego): typ, gromada lub klasa, rząd, rodzina, rodzaj, gatunek, podgatunek, odmiana; według powszechnie przyjętych poglądów takson może obejmować tylko formy pochodzące od jednego gatunku macierzystego (monofiletyzm)


