Przeczytaj
Enzymy restrykcyjne
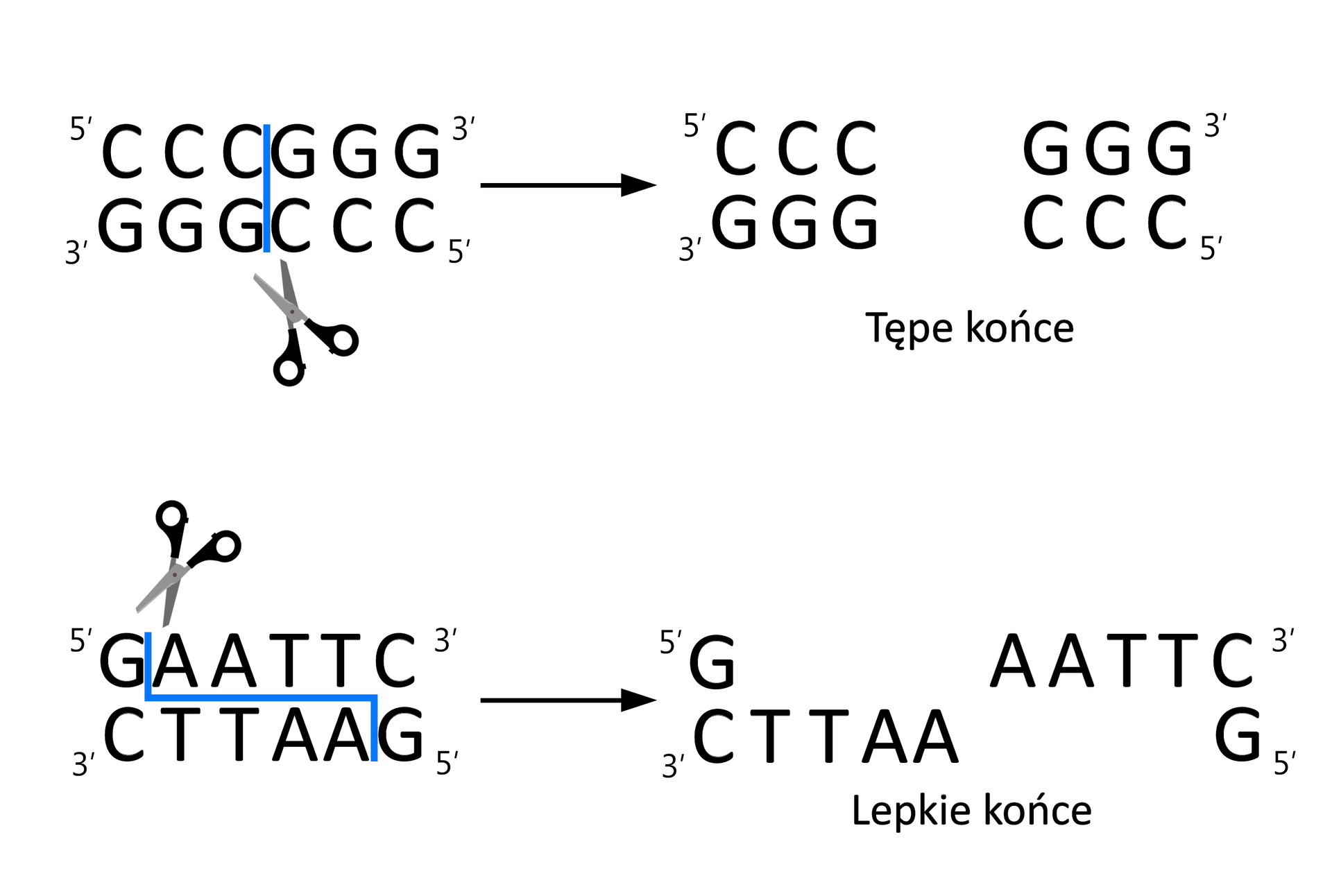
Enzymy restrykcyjne (restryktazy) są enzymami należącymi do endonukleazendonukleaz. Rozpoznają one specyficzne sekwencje DNA długości od kilku do kilkunastu nukleotydów (zwykle 4–8 pz). Zazwyczaj są to sekwencje palindromowe, czyli takie same, gdy odczytuje się je na obu niciach w kierunku od 5′ do 3′. Rozpoznanie sekwencji charakterystycznej dla danego enzymu powoduje hydrolizę wiązań fosfodiestrowych, najczęściej w obrębie sekwencji rozpoznawanej przez enzym. Powstałe fragmenty restrykcyjne DNA mogą być zakończone lepkimilepkimi bądź tępymitępymi końcami, w zależności od rodzaju enzymu. Duża liczba różnych enzymów restrykcyjnych oraz ich specyficzność w rozpoznawaniu określonych sekwencji DNA pozwalają na wykorzystanie ich działania w procesach inżynierii genetycznej.
Znanych jest ponad 1000 enzymów restrykcyjnych. Nazwy restryktaz wykorzystywanych w laboratoriach składają się z dwóch lub trzech członów. Pierwszy z nich zawiera trzyliterowy symbol informujący o gatunku bakterii, z której enzym został wyizolowany. Kolejny człon, czyli dodatkowa wielka litera, oznacza szczep bakterii, natomiast ostatni człon to cyfra rzymska określająca serię, np.:
EcoRI – pierwszy enzym wyizolowany z Escherichia coli szczepu R;
BamHI – pierwszy enzym wyizolowany z Bacillus amyloliquefaciens szczepu H.
Enzymy restrykcyjne dzieli się na kilka klas, ze względu na miejsce cięcia sekwencji DNA oraz niezbędne kofaktorykofaktory:
klasa I - dokonują cięcia w odległości nawet 1000 pz (par zasad) od rozpoznawanej sekwencji; kofaktorami w reakcji są m. in. ATP, jony magnezu (MgIndeks górny 2+2+) oraz S‑adenozylometioninaS‑adenozylometionina;
klasa II - przecinają dwuniciowy DNA w obrębie rozpoznawanej sekwencji palindromowej; kofaktorem enzymów tej klasy są jony magnezu (MgIndeks górny 2+2+);
klasa III - hydrolizują wiązania w tylko jednej nici DNA w odległości do 25 nukleotydów od specyficznej sekwencji; wymagane kofaktory to ATP i jony magnezu (MgIndeks górny 2+2+); S‑adenozylometionina wzmacnia aktywność enzymów tej klasy.
Najczęściej używanymi enzymami restrykcyjnymi w biologii molekularnej są enzymy klasy II, ze względu na fakt przecinania DNA w obrębie rozpoznawanej przez nie sekwencji palindromowej. Dzięki temu możliwe jest obliczenie wielkości powstałego po cięciu produktu i jego identyfikacja.
Więcej informacji o enzymach restrykcyjnych znajdziesz w materiale: Enzymy stosowane w biotechnologii molekularnejEnzymy stosowane w biotechnologii molekularnej.
Wykorzystanie enzymów restrykcyjnych
Dzięki obecności miejsc restrykcyjnych możliwe jest np. wprowadzanie do wektorawektora genów pochodzących od innych organizmów na drodze rekombinacji homologicznejrekombinacji homologicznej. Proces ten opiera się na działaniu restryktaz dokonujących cięcia DNA oraz ligazy, która łączy wolne końce powstałych cząsteczek. Znajomość specyficzności danego enzymu restrykcyjnego pozwala na zaprojektowanie fragmentów DNA w taki sposób, aby możliwe było połączenie wstawkiwstawki z wektorem (np. plazmidem).
Aby ułatwić wprowadzanie modyfikacji genetycznych, tworzone są mapy restrykcyjne plazmidów, które wskazują ułożenie miejsc na cząsteczce ciętych przez różne enzymy restrykcyjne. Dzięki nim planując doświadczenie, można dobrać enzym, który rozpoznaje sekwencje obecne w plazmidzie oraz określić, w jakim miejscu plazmid zostanie przecięty.

Aby wprowadzić do plazmidu pożądane DNA należy zastosować te same enzymy, których użyto do trawienia wstawki. Dzięki obecności lepkich końców o takiej samej sekwencji w obu fragmentach możliwe jest połączenie ich za pomocą ligazy DNA. Następnie plazmidy można namnożyć w komórkach bakteryjnych w celu uzyskania większej ilości kopii.
Wydajność ligacji jest o wiele większa w przypadku lepkich końców, ponieważ mogą one łączyć się wiązaniami wodorowymi z komplementarnymi jednoniciowymi końcami innych cząsteczek DNA.
Aby gen ze wstawki ulegał ekspresji, musi ona zostać wbudowana w odpowiedniej orientacji, tak aby sekwencja genu znajdowała się za promotorem bakteryjnym, w orientacji 5′–3′ na nici kodującej i kończyła sygnałem prowadzącym do terminacji transkrypcji. Jednak ponieważ lepkie końce na obu stronach wstawki są takie same, dlatego może ona zostać połączona z plazmidem w odwrotnej orientacji do zamierzonej. W takim przypadku sekwencja genu byłaby odczytywana „od tyłu”, przez co nie uległby on ekspresji.
Więcej o plazmidach znajdziesz w e‑materiałach:
Wektory genetyczneWektory genetyczne;
Klonowanie DNAKlonowanie DNA.
Analiza restrykcyjna
Aby sprawdzić, czy zaszła ligacja wstawianego genu z plazmidem, oraz w jakiej orientacji został wstawiony gen, dokonywana jest analiza restrykcyjna. Technika ta pozwala na identyfikację fragmentów powstających po cięciu enzymem restrykcyjnym na podstawie ich wielkości.
Analizę restrykcyjną przeprowadza się poprzez poddanie plazmidów trawieniu restrykcyjnemu. Trawienie restrykcyjne zachodzi w środowisku wodnym z buforem, który zapewnia niezbędne warunki reakcji. Czas reakcji zależy od rodzaju enzymu i wielkości materiału poddawanego trawieniu. Użyte w reakcji enzymy powinny trawić wstawkę, a następnie plazmid. Po trawieniu przeprowadza się ligację wstawki z plazmidem. Na podstawie długości powstałych po trawieniu fragmentów można odróżnić plazmid zawierający wstawkę od tego, który nie został połączony z żadnym fragmentem DNA.
Kolejnym krokiem w analizie restrykcyjnej jest wizualizacja produktów trawienia. Dokonuje się jej na drodze elektroforezy.
Elektroforeza DNA
Elektroforeza to metoda polegająca na poruszaniu się naładowanych cząstek pod wpływem pola elektrycznego. Cząstki mające na swojej powierzchni ładunek dodatni dążą do elektrody ujemnej (katody), mające zaś ładunek ujemny – do elektrody dodatniej (anody).
W DNA obecne są grupy fosforanowe, przez co ma on ładunek ujemny. Dzięki temu wszystkie cząsteczki tego kwasu nukleinowego migrują w stronę dodatniego bieguna pola elektrycznego, a szybkość migracji zależy od wielkości cząsteczek – im mniejsza (krótsza) cząsteczka DNA lub RNA, tym szybciej przesuwa się w stronę elektrody dodatniej.
Elektroforeza żelowa pozwala na rozdzielenie kwasów nukleinowych w porowatym żelu. Żel to galaretowata substancja, która składa się z agarozy lub poliakrylamidu. Wylewa się go cienką warstwą do szklanego pojemnika. Działa on jak sito, umożliwiając separację cząsteczek. Są w nim wydrążone studzienki, w których umieszcza się próbki, np. zawierające plazmidy po trawieniu restrykcyjnym. Oprócz badanych próbek na żelu umieszcza się również mieszaninę fragmentów DNA o znanej wielkości, tzw. marker masy nazywany też drabinką (ang. ladder).
Żel umieszcza się w aparacie do elektroforezy wypełnionym buforem. Następnie poddaje się go działaniu pola elektrycznego. Pod jego wpływem fragmenty kwasów nukleinowych o różnej wielkości przemieszczają się od katody w stronę anody z różną szybkością. Mniejsze cząsteczki migrują szybciej, gdyż łatwiej przeciskają się w sieci żelu, natomiast cząsteczki, które są większe od porów, są prawie nieruchome. Dodatek odpowiedniego barwnika pozwala na obserwację tempa migracji próbek w żelu. Elektroforeza prowadzona jest pod napięciem 100–120 V przez ok. 20–30 min.
Po zakończonej elektroforezie żel analizuje się w świetle UV. Rozdzielone fragmenty widoczne są w postaci prążków dzięki świeceniu barwników fluorescencyjnychfluorescencyjnych wiążących się z DNA.
Liczba prążków świadczy o tym, ile miejsc restrykcyjnych zostało rozpoznanych przez enzymy restrykcyjne w danej cząsteczce DNA.
Wielkość fragmentów DNA określa się na podstawie porównania ich szybkości migracji w żelu (na podstawie pozycji odpowiednich prążków) z szybkością migracji fragmentów DNA o znanej długości (pozycje prążków z drabinki).
Elektroforezę DNA stosuje się m.in. w biologii molekularnej, farmakologii, medycynie sądowej, weterynarii, diagnostyce medycznej oraz kontroli jakości żywności. Wykorzystywana może być także w kryminalistyce do identyfikowania osób podejrzanych o popełnienie przestępstwa. Wówczas próbkę zebraną z miejsca przestępstwa oraz próbki uzyskane od osób podejrzanych nakłada się na żel. Po zakończonej elektroforezie należy je porównać. Jeśli układ prążków w danej próbce jest taki sam jak w próbce pobranej z miejsca przestępstwa, dana osoba najprawdopodobniej jest winna.
Słownik
jedno ze zjawisk elektrokinetycznych polegające na poruszaniu się naładowanych cząsteczek pod wpływem pola elektrycznego; cząsteczki mające na swojej powierzchni ładunek dodatni dążą do elektrody ujemnej, natomiast obdarzone ładunkiem ujemnym – do elektrody dodatniej; jest to także technika rozdziału cząsteczek pod wpływem pola elektrycznego dzięki różnicom w masie i/lub ładunku
grupa enzymów należąca do hydrolaz – enzymów katalizujących cięcie cząsteczek kwasów nukleinowych
zjawisko polegające na emitowaniu światła przez wzbudzony atom lub cząsteczkę, spowodowane wzbudzeniem przez pochłonięcie światła lub innego promieniowania elektromagnetycznego
grupa związków chemicznych potrzebnych enzymom do katalizowania reakcji chemicznych; kofaktory nieorganiczne to m.in. jony metali; kofaktory organiczne to np. witaminy
powstające po trawieniu restrykcyjnym jednoniciowe końce dwuniciowych cząsteczek DNA, które są komplementarne do innego jednoniciowego odcinka na końcu drugiej cząsteczki DNA
procesy prowadzące do powstania nowych genotypów, w wyniku niezależnej segregacji genów leżących na różnych chromosomach oraz zmiany w obrębie nici DNA
pochodna adenozyny i metioniny, u człowieka jest głównym substratem w procesach metylacji, czyli przyłączania grup metylowych
powstające po trawieniu restrykcyjnym dwuniciowe zakończenie fragmentu DNA
niewielka cząsteczka DNA zdolna do replikacji w komórce biorcy; podstawowe narzędzie inżynierii genetycznej; może zostać zrekombinowana przez włącznie do niej fragmentu innej cząsteczki DNA, który po wprowadzeniu do komórek gospodarza będzie w nich powielany i poddawany ekspresji; do komórek ssaków wektory można wprowadzać za pośrednictwem wirusów
fragment DNA włączony np. do plazmidu dzięki technikom inżynierii genetycznej